8月23日,國際學術期刊Cell Reports在線發表了中國科學院分子細胞科學卓越創新中心(生物化學與細胞生物學研究所)石建濤研究員與上海交通大學醫學院附屬瑞金醫院方海教授的最新研究成果“Toward the DNA methylation haplotype map of 11 common solid cancers”。該研究通過全基因組亞硫酸鹽測序(WGBS),繪制了11種中國人群常見實體瘤的DNA甲基化單體圖譜(DNA methylation haplotype map),并鑒定出81,567個DNA甲基化單體水平上的共甲基化區間,稱為DNA甲基化單體塊(Methylation Haplotype Blocks,MHBs)。大規模數據整合分析表明,腫瘤MHB富集了腫瘤特異性調控元件,和腫瘤細胞的基因表達調控顯著相關,也能作為腫瘤液體活檢的標志物。
DNA甲基化是重要的表觀遺傳修飾,在基因表達調控中發揮關鍵作用。傳統研究策略通常采用CpG位點甲基化平均水平作為評估標準,據此劃分出無甲基化區域(UMR)、低甲基化區域(LMR)以及部分甲基化結構域(PMD)等功能區塊(Burger et al.,2013)。然而,這種基于均值的分析方法存在重要缺陷:僅考慮甲基化的整體水平,忽略了單個DNA分子上的甲基化分布模式,即DNA甲基化單體特征。相同的平均甲基化水平可能對應不同的單體甲基化構型,產生截然不同的轉錄調控效果。不僅如此,鄰近CpG位點的甲基化狀態并非隨機分布,而是可能存在相關性。受此啟發,研究者運用群體遺傳學的連鎖分析理念,識別出具有協同甲基化特性的基因組片段,并將其命名為甲基化單體塊(MHB)(Shoemaker et al.,2010;Guo et al.,2017)。這些區域被認為是結構穩定的基因組單元,在胚胎發育過程中維持相對穩定。盡管如此,MHB在不同腫瘤中的分布特征及其潛在的生物學意義,仍需深入研究。
在以往的研究中,石建濤研究組創建了新的DNA甲基化單體格式mHap(Zhang et al.,2021),并開發了配套分析軟件mHapTk(Ding et al.,2022)和數據平臺mHapBrowser(Hong et al.,2023),為大規模整合分析提供了技術支撐。該團隊與方海研究組合作,利用海量公共WGBS數據資源,克服了傳統"甲基化均值"分析的瓶頸,構建了涵蓋17種正常組織的DNA甲基化單體圖譜(Feng et al. 2023),并發現了一類由甲基化單體特征定義的轉錄調控元件。
在此基礎上,兩個研究組再度合作,將研究目標轉向高度異質性的實體腫瘤。首次在泛癌水平上對中國人群11種常見、原發實體瘤樣本進行深度WGBS測序,共鑒定出 81,567個非冗余的腫瘤MHB。這些基因組區間富集了腫瘤特異性調控元件,在排除了差異甲基化區間的影響后,仍然同腫瘤差異表達的基因顯著相關,并在多個腫瘤類型中富集G2/M檢查點、MYC靶基因和E2F信號通路等致癌通路。腫瘤MHB可用于分析腫瘤DNA甲基化異質性,發現甲基化失調與驅動性突變存在關聯,且富集于炎癥相關通絡。在轉化應用方面,腫瘤MHB可為復雜癌癥的早期預警和分子檢測提供高度特異性的潛在標志物。綜上所述,腫瘤MHB作為多模態表觀遺傳調控因子,有效連接了腫瘤異質性、轉錄調控和液體活檢診斷,為腫瘤復雜生物學特征解析及臨床診斷應用提供了新途徑。
分子細胞卓越中心研究生洪毓陽以及上海交通大學張志強博士為該論文的共同第一作者。分子細胞卓越中心石建濤研究員和上海交通大學醫學院附屬瑞金醫院方海教授為該論文的共同通訊作者。該項研究得到了分子細胞卓越中心高性能計算平臺的大力支持。該項目由國家自然科學基金資助。
文章鏈接:https://www.sciencedirect.com/science/article/pii/S2211124725009684
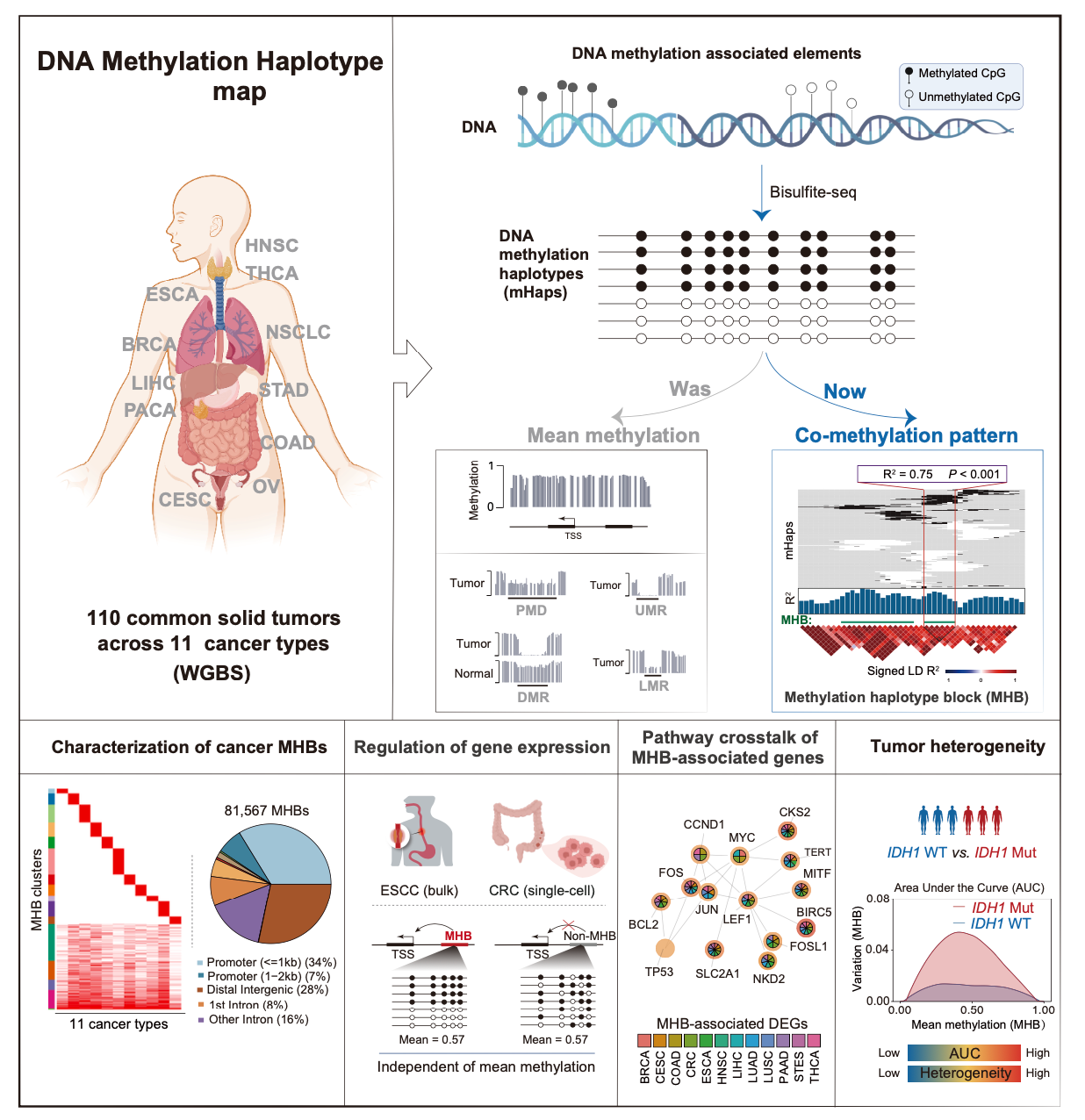
腫瘤DNA甲基化單體圖譜

 日韩欧美精品三级,激情婷婷亚洲,日韩午夜一区,夜夜夜精品看看
主站蜘蛛池模板:
综合精品久久|
在线看国产一区二区|
日韩欧美中文一区|
成人在线电影在线观看视频|
欧美日韩精品在线观看|
欧美日韩精品国产|
一本到12不卡视频在线dvd|
日韩电影中文字幕在线观看|
日韩成人精品一区|
久久免费视频这里只有精品|
国产丝袜一区二区三区|
国产视频久久网|
中文字幕一区二区三区视频|
欧美蜜桃一区二区三区|
国产精品亚洲二区|
欧美日韩性生活视频|
日韩欧美综合|
欧美色网站导航|
久久蜜桃av一区二区天堂|
国产精品国产精品国产专区不蜜|
久久久国产精品一区|
久久中文字幕在线视频|
残酷重口调教一区二区|
94色蜜桃网一区二区三区|
日本不卡二区|
久久精品亚洲乱码伦伦中文|
久久麻豆精品|
欧美日韩一区精品|
中文字幕亚洲国产|
亚洲小视频在线观看|
婷婷久久国产对白刺激五月99|
一本大道久久a久久精品综合|
国产三级一区二区|
国产一区二区不卡|
色呦呦国产精品|
久久电影网站中文字幕|
日韩中文第一页|
亚洲精品国产精品国自产观看|
久久久国产精品免费|
午夜精品久久久久久久99樱桃|
美女亚洲一区|
日韩欧美精品三级,激情婷婷亚洲,日韩午夜一区,夜夜夜精品看看
主站蜘蛛池模板:
综合精品久久|
在线看国产一区二区|
日韩欧美中文一区|
成人在线电影在线观看视频|
欧美日韩精品在线观看|
欧美日韩精品国产|
一本到12不卡视频在线dvd|
日韩电影中文字幕在线观看|
日韩成人精品一区|
久久免费视频这里只有精品|
国产丝袜一区二区三区|
国产视频久久网|
中文字幕一区二区三区视频|
欧美蜜桃一区二区三区|
国产精品亚洲二区|
欧美日韩性生活视频|
日韩欧美综合|
欧美色网站导航|
久久蜜桃av一区二区天堂|
国产精品国产精品国产专区不蜜|
久久久国产精品一区|
久久中文字幕在线视频|
残酷重口调教一区二区|
94色蜜桃网一区二区三区|
日本不卡二区|
久久精品亚洲乱码伦伦中文|
久久麻豆精品|
欧美日韩一区精品|
中文字幕亚洲国产|
亚洲小视频在线观看|
婷婷久久国产对白刺激五月99|
一本大道久久a久久精品综合|
国产三级一区二区|
国产一区二区不卡|
色呦呦国产精品|
久久电影网站中文字幕|
日韩中文第一页|
亚洲精品国产精品国自产观看|
久久久国产精品免费|
午夜精品久久久久久久99樱桃|
美女亚洲一区|